Heterochromatin – Verpackungskünstler am Werk
Forschungsbericht (importiert) 2012 - Max-Planck-Institut für Immunbiologie und Epigenetik
Epigenetik: Genkontrolle durch Genomverpackung
Sind wir mehr als die Summe unserer Gene und wie können Umwelteinflüsse die Genexpression verändern? Antworten auf diese Fragen bietet eine neue zukunftsweisende Forschungsrichtung, die Epigenetik. Im Gegensatz zur Genetik beschäftigt sich die Epigenetik mit Ausprägungen, die nicht durch die DNA-Sequenz bedingt sind (Epigenetik = zusätzlich zur DNA-Sequenz). (Fast) alle Zelltypen (ca. 200) des Menschen besitzen zwar die gleiche genetische Information, verhalten sich jedoch sehr unterschiedlich in ihrer Umsetzung. So kann zum Beispiel eine Stammzelle nicht allein aufgrund der DNA-Sequenz erkannt werden und nur eines der beiden X-Chromosomen in weiblichen Zellkernen ist aktiv. Der DNA-Faden liegt nicht nackt im Zellkern vor, sondern ist über Proteinkugeln (Histone) verpackt und geschützt. Dieses DNA-Histon-Polymer wird Chromatin genannt. Ein hoher Verpackungsgrad (geschlossenes Chromatin oder Heterochromatin) legt Gene still, wogegen ein geringer Verpackungsgrad (offenes Chromatin oder Euchromatin) Genaktivität ermöglicht. Epigenetische Veränderungen, wie z. B. chemische Modifizierungen der Histone oder der DNA, erlauben plastische Übergänge zwischen diesen beiden Chromatinzuständen und somit die Herstellung einer Vielzahl epigenetischer Varianten unseres Genoms (Epigenom) (Abb. 1).

Euchromatin und Heterochromatin: die zwei Gesichter des Chromatins
Basierend auf ihrem Verpackungsgrad kann man zwei Grundtypen des Chromatins definieren: Euchromatin und Heterochromatin. Im Euchromatin findet sich die Mehrzahl der Gene. Es trägt viele aktivierende Modifizierungen und ist somit relativ leicht zugänglich für die Transkriptionsmaschinierie. Im Gegensatz dazu enthält das Heterochromatin hauptsächlich repetitive Sequenzen, die nicht-kodierende RNA-Moleküle bilden können. Um die Transkription dieser Regionen gering zu halten, wird das Heterochromatin durch repressive chemische Modifizierungen dicht verpackt. Heterochromatin-Abschnitte liegen beispielsweise rund um die Zentromere und an den Chromosom-Enden, den Telomeren. Die Etablierung und Aufrechterhaltung dieses konstitutiven Heterochromatins ist äußerst wichtig für Zelltyp-Identität, Genregulation und eine korrekte Chromosomensegregation (Abb. 2).

Thomas Jenuwein und sein Team arbeiten seit zwei Dekaden an den molekularen epigenetischen Mechanismen, die die Bildung von Heterochromatin steuern. Im Jahr 2000 gelang der Forschungsgruppe mit der Identifizierung der ersten Histonmethyltransferase (Suv39h) eine bahnbrechende Entdeckung [2]. Diese Klasse von Enzymen versieht Histone an unterschiedlichen Stellen mit Methyl-Gruppen und beeinflusst dadurch die Chromatinstruktur. Neben den Suv39h-Enzymen kennt man zu diesem Zeitpunkt bis zu 14 weitere Kernkomponenten des Heterochromatins, die sowohl weitere Enzyme als auch strukturelle Proteine einschließen [3]. Dennoch bleiben viele offene Fragen, z. B.: Gibt es weitere Komponenten des Heterochromatins, was unterscheidet Euchromatin von Heterochromatin und welche Rolle spielen die nicht-kodierenden RNAs, die von den repetitiven Sequenzen gebildet werden? Im Folgenden werden zwei neue Studien der Arbeitsgruppe von Thomas Jenuwein zusammengefasst, die Antworten auf einige dieser Fragen liefern.
Eins, zwei oder drei: Lysin-Methylierung am Heterochromatin
Ines Pinheiro, Doktorandin im Jenuwein-Labor, hat zwei weitere Methyltransferasen entdeckt, die eine wichtige Funktion für die Aufrechterhaltung des Heterochromatins haben [4]. Prdm3 und Prdm16 sind Methyltransferasen, die an das Histon H3 in der Position Lysin 9 (H3K9) eine Methylgruppe anheften. Die beiden Enzyme waren bislang nur als Transkriptionsfaktoren bekannt, die die Aktivität verschiedener Gene regulieren. Wie wichtig Prdm3 und Prdm16 sind, zeigen Experimente, in denen beide Enzyme ausgeschaltet wurden. Das Heterochromatin bricht danach zusammen und die heterochromatischen Regionen können abgelesen werden (Abb. 3).

Dieser Zerfall des Heterochromatins erklärt sich aus einem sequentiellen Mechanismus für die Histonmethylierung am Heterochromatin: Prdm3 und Prdm16 heften eine Methyl-Gruppe an H3K9. Dieses einfach methylierte H3 (H3K9me1) wird in den Zellkern transportiert und in das Heterochromatin eingebaut. An das einfach methylierte Histon können dann andere Methyltransferasen wie Suv39h noch zwei weitere Methyl-Reste (H3K9me3) anfügen und das Heterochromatin so weiter stabilisieren. Die Freiburger Forscher beobachteten zudem, dass ohne Prdm3 und Prdm16 die Lamina des Zellkerns zerstört wird. Das Heterochromatin muss mit dieser Schicht aus Lamina-Proteinen unterhalb der Kernhülle verbunden sein. Die Ursache für die Beeinträchtigung der Lamina ist im Moment allerdings noch nicht geklärt. Es ist denkbar, dass dafür der Verlust des Heterochromatins oder auch die fehlende Methylierung eines Lamina-Proteins verantwortlich ist. Beide Hypothesen sind gegenwärtig Gegenstand von Forschungsprojekten.
Transkriptionsfaktoren: Berge und Täler im Heterochromatin
Aber nicht nur die Methylierung von Histonen ist für die Aufrechterhaltung von Heterochromatin-Regionen erforderlich. In einer weiteren Studie haben die Doktorandinnen Aydan Karslioglu und Valentina Perrera die Rolle von Transkriptionsfaktoren untersucht, also Proteinen, die an die DNA binden und die Aktivität von Genen kontrollieren – im Falle des Heterochromatins die Unterdrückung von nicht-kodierenden RNA-Molekülen [5]. Zwei davon sind demzufolge für ein intaktes Heterochromatin unverzichtbar: Pax3 und Pax9. Nur wenn diese beiden Transkriptionsfaktoren und ihre Bindungsstellen in der repetitiven DNA vorhanden sind, bleibt das Heterochromatin bestehen. Die Forscher gehen jedoch davon aus, dass auch noch weitere Transkriptionsfaktoren an repetitive Sequenzen im Heterochromatin binden können.
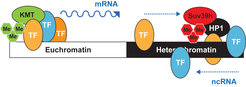
Transkriptionsfaktoren kontrollieren also die Genaktivität sowohl im Euchromatin als auch im Heterochromatin. Trotzdem gibt es Unterschiede zwischen den beiden: Im Heterochromatin sind die Bindungsstellen relativ zufällig über den DNA-Strang verteilt, im Euchromatin dagegen häufen sie sich an den für die Genregulation wichtigen Stellen. Man könnte die Verteilung im Heterochromatin mit den Aiguilles Droites im Mont-Blanc-Massiv vergleichen: Viele kleine Gipfel ohne tiefe Täler dazwischen. Das Euchromatin gleicht eher dem Matterhorn: Ein hoher Gipfel ohne Nebengipfel. Ein wichtiger Unterschied zwischen Heterochromatin und Euchromatin liegt für die Forscher in der Kontrolle der Genaktivität und der Bildung von RNAs. Im Heterochromatin sind die Bindungsstellen für Transkriptionsfaktoren eher zufällig verteilt, so dass sie sich nicht synergistisch verstärken können. Die DNA kann deshalb dort nicht so abgestimmt abgelesen werden. Insgesamt überwiegen hemmende Einflüsse, die das Heterochromatin weitgehend abschalten. Im Euchromatin dagegen binden die Transkriptionsfaktoren so an die DNA, dass sie sich ergänzen. Dies erlaubt eine fein abgestimmte Kontrolle der Genaktivität (Abb. 4).



