Mediator der transkriptionellen Regulation
Forschungsbericht (importiert) 2006 - Max-Planck-Institut für Immunbiologie und Epigenetik
Einleitung
Fast alle Lebensvorgänge werden von Proteinen gesteuert. Auf den Chromosomen ist die Protein-Bauanleitung in Form von Genen gespeichert. Wird ein Protein benötigt, wird es gezielt von der RNA-Polymerase II abgelesen. Bei diesem Kopiervorgang, den man auch Transkription nennt, entsteht zunächst mRNA (messenger- oder Boten-RNA). Gleichsam als Bote zwischen Zellkern und Ribosom fungiert die mRNA als mobile DNA-Kopie. Am Ribosom wird sie dann in Aminosäuren (die Primärstruktur eines Proteins) übersetzt beziehungsweise translatiert.
Die Regulation der Transkription von Genen ist von zentraler Bedeutung für die Zelldifferenzierung. Eine Fehlregulation kann zu Krankheiten wie Krebs führen. Das spezifische Ablesen von Genen bestimmt die Identität von z.B. einer Blutzelle oder einer Riechzelle. Transkriptionelle Regulation findet vor allem beim Beginn, der Initiation oder dem Kettenstart, statt. Hierfür sind zusätzliche Faktoren notwendig, die nach Einsetzen der RNA-Synthese nicht mehr gebraucht werden. Diese zusätzlichen Faktoren können in allgemeine und genspezifische Faktoren unterteilt werden. Die allgemeinen Transkriptionsfaktoren (TFIIB, -D, -E, F und -H) können zusammen mit der RNA-Polymerase II den Minimal-Promotor, den Startpunkt der Transkription, erkennen, wohingegen die Aktivatoren/Repressoren an genspezifische Promoter-Elemente binden. Transkriptionelle Regulation ist die Kommunikation zwischen genspezifischen und allgemeinen Transkriptionsfaktoren.
Die Rolle des Mediatorkomplexes in der transkriptionellen Regulation
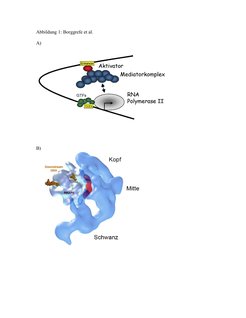
Der Mediator übersetzt genspezifische Signale und gibt diese an die allgemeine Transkriptionsmaschinerie weiter, indem er physisch mit spezifischen Aktivatoren und der RNA-Polymerase II wechselwirken kann (Abb. 1A). Der Mediator wurde vor fünfzehn Jahren im Labor von Roger Kornberg – diesjähriger Nobelpreisträger für Chemie – als Aktivität entdeckt, die für die transkriptionelle Aktivierung im zellfreien System benötigt wird [1, 2 und 3]. Der Mediator kann mit der RNA-Polymerase an drei Stellen interagieren [4]; siehe Abbildung 1B. Der Mediatorkomplex besteht aus 28 Untereinheiten, die in vier Module gegliedert werden können [3; 5]. Funktionell kontrolliert der Mediatorkomplex die Phosphorylierung der C-terminalen Domäne der größten Untereinheit der RNA-Polymerase II. Diese Phosphorylierung der RNA-Polymerase II ist von zentraler Bedeutung für die Regulation der Transkription [2; 3].
Durch genaue Quantifikationsmethoden stellten Tilman Borggrefe und sein Team fest, dass der Mediatorkomplex eine limitierende Komponente in der Transkriptionsmaschinerie ist [6]. In Chromatin-Immunopräzipitations-Experimenten konnten sie zeigen, dass der Mediator von transkriptionellen Aktivatoren unabhängig von der RNA-Polymerase rekrutiert werden kann [7].
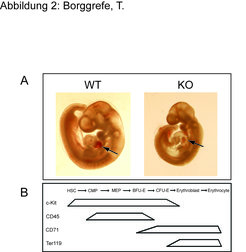
Die oben genannten Experimente beschreiben den molekularen Mechanismus, weniger die physiologische Relevanz des Mediatorkomplexes. Mithilfe von Knock-out-Mäusen konnten die Forscher erste Einblicke in die physiologische Bedeutung des Mediatorkomplexes gewinnen. Bei der Analyse von Mausembryonen, denen die Mediatoruntereinheit Med1 fehlt, konnten sie feststellen, dass die Embryonen anämisch sind (Abb. 2A). Aufgrund dieses Befundes untersuchten sie die Entwicklung der roten Blutzellen im Detail und klärten den molekularen Mechanismus auf [8].
Erythropoese als ein hervorragendes experimentelles System
Erythropoese – die Entwicklung von einer Stammzelle zu roten Blutkörperchen – ist ein sehr gut verstandenes System, weil es zum einen durch seine klinische Relevanz schon lange untersucht wurde, zum anderen weil Blutzellen leicht zugänglich sind. Mithilfe von Wachstumsfaktoren wie Erythropoietin, die klinisch unter anderem bei Anämie-Patienten eingesetzt werden, kann man so genannte Kolonie-Assays durchführen. Das heißt, man plattiert Stammzellen in einem viskosen Medium aus, sodass diese sich nicht mehr bewegen können, Faktoren jedoch frei diffundieren können. Nach ein paar Tagen färbt man alle gewachsenen Zellkolonien mit einem Farbstoff an, der spezifisch an Hämoglobin bindet, und zählt die blau gefärbten Kolonien (Abb. 3A). Diese hämatopoetischen Kolonie-Assays wurden bereits in den 70er-Jahren durchgeführt und zahlreiche Krankheiten wie Anämien und Leukämien damit charakterisiert. Die Wissenschaftler am MPI für Immunbiologie setzten diese Assays ein, um die Funktion von Mediator-defizienten Vorläuferzellen zu überprüfen. Mithilfe von charakteristischen Oberflächenproteinen konnten sie erythroide Vorläufer sortieren und anschließend Kolonie-Assays durchführen (siehe Abb. 3A,C und Abb. 2B).
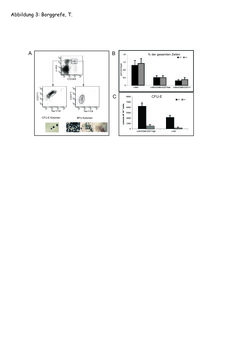
Zu Beginn der 90er-Jahre ist es möglich geworden, Gene spezifisch auszuschalten. Gen-defiziente oder Knock-out-Mäuse haben die medizinische Forschung entscheidend vorangebracht, weil man ein Gen nun unter definierten genetischen Bedingungen genau unter die Lupe nehmen konnte. In der Mediator-defizienten Maus sind z.B. die hämatopoetischen Stammzellen zwar noch vorhanden, aber die Koloniebildung von erythroiden Vorläuferzellen ist extrem reduziert (Abb. 3C). Dieser Phänotyp ähnelt dem von Mäusen, denen der erythrospezifische Schlüsseltranskriptionsfaktor GATA-1 fehlt.
Der Mediatorkomplex ist ein entscheidender Kofaktor für den erythrospezifischen Transkriptionsfaktor GATA-1
Das Gen für den Trankriptionsfaktor GATA-1 wird sehr spezifisch erst in den späteren Stadien der Erythropoese angeschaltet. Mäuse, die kein GATA-1-Gen mehr haben, sterben schon in der Mitte der embryonalen Entwicklung an Anämie. GATA-1 ist nicht nur für die Expression von Hämoglobingenen essentiell, sondern für fast alle erythrospezifischen Zielgene. Damit ist es ein Schlüsseltranskriptionsfaktor oder “Master regulator”.
Da der Phänotyp der Mediator-defizienten Mäuse dem der GATA-1-defizienten Mäuse ähnelt, untersuchten die Wissenschaftler in Freiburg, ob der Mediator ein essentieller Kofaktor für GATA-1 ist. Hierfür verwendeten sie die oben genannten Kolonie-Assays. Nach 3 Tagen Kultur mit dem Wachstumsfaktor Erythropoetin bilden sich kompakte Kolonien aus 16 bis 32 Zellen, die man auch CFU-Es (Colony forming units-erythrocyte) nennt. Stammzellen entwickeln sich zunächst in CFU-E-Vorläufer und dann anschließend zu reifen Erythrozyten (siehe Abb. 2B). Mediator-defiziente Vorläufer haben eine normale Anzahl an Stammzellen, zeigen aber drastisch weniger CFU-E-Koloniebildung (siehe Abb. 3C). In biochemischen Versuchen konnten die Forscher eine Protein-Protein-Wechselwirkung zwischen Med1 und GATA-1 nachweisen und in funktionellen Assays zeigen, dass Genaktivierung durch GATA-1 in Abwesenheit vom Mediator nicht mehr stattfindet. Sie schlussfolgern, dass der Mediator ein entscheidender Faktor in der Entwicklung von roten Blutkörperchen ist [8].
Ausblick
Diese Ergebnisse zeigen, dass der Mediatorkomplex, der ursprünglich in der Bäckerhefe entdeckt wurde, eine physiologische Relevanz in höheren Eukaryoten wie Maus und Mensch hat. Da die einzelnen Stadien der Erythropoese genau verstanden sind und innerhalb von Stunden die transkriptionelle Maschine für Globinproduktion angeschaltet werden kann, können die Wissenschaftler bald einen „molekularen Film” drehen, der zeigt, welche Faktoren zu welchem Zeitpunkt zu GATA-1-Zielgenen gelangen und welche Kofaktoren benötigt werden, damit die RNA-Polymerase II gezielt Gene ablesen kann.
Danksagung
Mein Dank geht an die Mitglieder meiner Arbeitsgruppe, vor allem Melanie Stumpf, und an meine Mentoren Roger Kornberg (Universität Stanford), Hans-Reimer Rodewald (Universität Ulm) und Rudolf Grosschedl (Max-Planck-Institut Freiburg) für ihre Unterstützung.
![A) Modell für die Bildung des RNA-Polymerase II-Initiationskomplexes: Mediator bindet an genspezifische Regulatoren und interagiert transient mit der RNA-Polymerase II. GTF=Generelle (allgemeine) Transkriptionsfaktoren. B) Elektronenmikroskopisch erstellte Struktur des Mediators (blau) im Komplex mit der RNA-Polymerase II (weiß); Darstellung mit Genehmigung aus [4].
A) Modell für die Bildung des RNA-Polymerase II-Initiationskomplexes: Mediator bindet an genspezifische Regulatoren und interagiert transient mit der](https://www.mpg.de/449608/original-1293749347.jpg?t=eyJ3aWR0aCI6MzQxLCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6NDQ5NjA4fQ%3D%3D--5d53962406ce1adf16c865b1648af9b4367d7b70)
![Mediator-defiziente Embryos sind anämisch:
A) Mediator-Untereinheit Med1/TRAP220-defiziente Embryos (KO) sind anämisch, nicht aber Wildtyp (WT)- Embryos. B) Spezifische Oberflächenmarker sind in der Differenzierung von Stammzellen zu Erythrozyten unterschiedlich exprimiert. HSC=Hämatopoetische Stammzelle, CMP=Common myeloid Progenitor, MEP=Myeloid/Erythroid Progenitor, BFU-E=Burst forming Unit erythrocyte, CFU-E=Colony forming unit erythrocyte; Darstellung mit Genehmigung aus [8]. Mediator-defiziente Embryos sind anämisch:
A) Mediator-Untereinheit Med1/TRAP220-defiziente Embryos (KO) sind anämisch, nicht aber Wildtyp (WT)- Embry](https://www.mpg.de/449664/original-1293750170.jpg?t=eyJ3aWR0aCI6MzQxLCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6NDQ5NjY0fQ%3D%3D--95afe9d635ad35bd9bbf252a2cd32fa97fad3602)
![Bildung von erythroiden Kolonien von Wildtyp- und Med1-defizienten Vorläuferzellen:
A) Vorläuferzellen wurden mit den Markern c-Kit, Ter-119 und CD71 sortiert und in Methylzellulose-Assays auf CFU-E- und BFU-E-Koloniebildung analysiert. B) Med1-defiziente- (KO) und Wildtyp- (Wt) Mäuse haben gleiche Mengen an c-Kit-positiven Vorläuferzellen. C) Med1-defiziente Vorläufer bilden 10x weniger CFU-E-Kolonien im Vergleich zum Wildtyp; Darstellung mit Genehmigung aus [8]. Bildung von erythroiden Kolonien von Wildtyp- und Med1-defizienten Vorläuferzellen:
A) Vorläuferzellen wurden mit den Markern c-Kit, Ter-119 und CD71](https://www.mpg.de/449720/original-1293749383.jpg?t=eyJ3aWR0aCI6MzQxLCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6NDQ5NzIwfQ%3D%3D--427fb1c8856eb4e67ff466e056c0271a5646efee)