Epigenetische Identität und Kernorganisation von Zentromeren
Forschungsbericht (importiert) 2013 - Max-Planck-Institut für Immunbiologie und Epigenetik
Einführung
Alle vererbbaren Zustände des Genoms, die nicht durch DNA selbst kodiert werden, nennt man epigenetisch. Dazu gehört die Regulation der Gen-Expression und es häufen sich Hinweise, dass bei den meisten Eukaryonten auch die Zentromer-Identität epigenetisch determiniert wird.
Das Zentromer spielt eine zentrale Rolle bei der korrekten Segregation der Chromosomen während der Zellteilung. Es wurde 1880 durch Walther Flemming als lichtmikroskopisch sichtbare, primäre Einschnürung definiert. Zentromere dienen als Plattform für den Aufbau eines Proteinkomplexes, dem sogenannten Kinetochor, der während der Zellteilung als Ansatzstelle für das Zellskeletts dient. Dies erlaubt, die beiden Schwesterchromosomen zu den gegenüberliegenden Zellpolen zu bewegen und gewährleistet so eine gleichmäßige Verteilung der genomischen Information auf beide Tochterzellen. Eine Fehlfunktion von Zentromeren hat gravierende Folgen für die betroffene Zelle und führt zu genomischer Instabilität und Aneuploidy, entweder durch asymmetrische Segregation von Chromosomen oder Chromosomenbruch bei Chromosomen mit mehr als einem Zentromer.
Zentromere sind epigenetisch determiniert
Wie die Position des Zentromers auf dem Chromosom bestimmt wird, ist eine langjährige Frage der Zellbiologie. Abgesehen von der Bäckerhefe, in der eine spezifische DNA-Sequenz das Zentromer „kodiert“, scheinen die meisten Organismen das Zentromer nicht durch die DNA, sondern epigenetisch zu identifizieren. Histon-Proteine binden DNA weitgehend Sequenz-unabhängig und helfen, das lange, fadenförmige DNA-Molekül zu verpacken. Ein besonders vielversprechender Kandidat für eine solche epigenetische Zentromer-Markierung ist deshalb eine Variante des Histon H3 namens CENP-AcenH3 (als CID in Drosophila bezeichnet), die als Teil des Nukleosoms ausschließlich DNA in Zentromer-Regionen bindet.
Überexpression des CENP-A Histons führt zu dessen Einbau in Chromosomenbereiche außerhalb des Zentromers und zur Bildung ektopischer Kinetochore [1]. Die neuen Kinetochore werden allerdings nicht zufällig, sondern bevorzugt am Übergang zwischen Gen-armen, inaktiven (Heterochromatin) und Gen-reichen, aktiven Abschnitten (Euchromatin) gebildet, oft nahe der Chromosomenenden, der sogenannten Telomere [2]. Für die Bildung eines Kinetochors ist neben den Zentromer-spezifischen Histonen also auch die Umgebung des Chromosom entscheidend. Dabei bleibt jedoch die Frage offen, ob CENP-A allein ausreicht, um die Bildung eines Zentromers auszulösen.
Mit einem biosynthetischen Ansatz in Zellkulturen der Fruchtfliege Drosophila melanogaster konnten wir den direkten Beweis für die epigenetische Natur des Zentromers liefern [3]. Dabei wurde dem CENP-A-Histon mithilfe einer künstlich angehängten DNA-Bindedomäne (dem Lac-Repressor; LacI) die Eigenschaft gegeben, direkt an eine spezifische, nicht-zentromerische DNA-Region, nämlich Lac Operatoren, zu binden. Dort werden dann funktionelle Kinetochore ausgebildet, die in der Mitose mit dem Zellskelett interagieren und erlauben, artifizielle Minichromosomen mit Lac Operatoren auf die beiden Tochterzellen zu verteilen. Das CENP-A-LacI Fusionsprotein ist zudem in der Lage, zelleigene CENP-A Proteine zu rekrutieren. Das ist eine essenzielle Eigenschaft, die den Fortbestand der epigenetischen Markierung über viele Zellgenerationen hinweg garantiert und erlaubt, Minichromosomen mit induzierten Zentromeren mehr als einen Monat lang stabil in der Drosophila Zellkultur zu halten (Abb. 1). Dies belegt, dass CENP-A eine Schlüsselrolle bei der epigenetischen Identität des Zentromers spielt und sowohl notwendig als auch ausreichend ist, um Zentromere zu bilden.
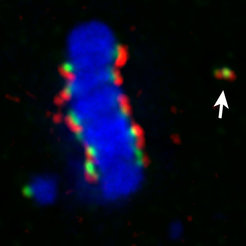
Nucleoplasmin bündelt und organisiert Zentromere im Zellkern
Zwei Meter Erbgut sind im Zellkern verpackt und eine wachsende Zahl von Untersuchungen weist darauf hin, dass die räumliche Organisation des Genoms im Zellkern nicht zufällig ist. Vor über 30 Jahren konnte bereits in Purkinje-Nervenzellen aus dem Gehirn der Maus beobachtet werden, dass Zentromere in Gruppen gebündelt sind. Solche Zentromer-Cluster assoziieren oft mit dem größten Subkompartiment des Zellkerns, dem Nukleolus, der hauptsächlich für die Bildung der Ribosomen verantwortlich ist. Diese spezifische Zellkernorganisation ist sowohl vom Zelltyp als auch vom Zellzyklus abhängig und konnte inzwischen auch in der Fruchtfliege und in menschlichen Zellen nachgewiesen werden (Abb. 2). Allerdings konnten bis jetzt weder die Proteine noch die Mechanismen identifiziert werden, die an dieser speziellen Zellkernorganisation beteiligt sind. Ein interessanter Kandidat dafür ist das Protein Nucleophosmin (NPM1), das vorwiegend im Nukleolus lokalisiert ist und in menschlichen Zellen mit CENP-A interagiert [4]. Wir konnten kürzlich zeigen, dass das Drosophila Homolog von NPM1, Nucleoplasmin (NLP), tatsächlich für die Organisation von Zentromeren in Zellen der Fruchtfliege verantwortlich ist [5].
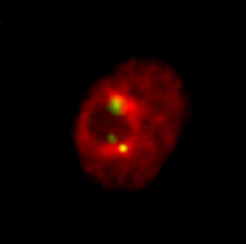
Wenn NLP mit der sogenannten Knockdown-Methode (RNAi) aus den Zellen entfernt wird, fallen die gebündelten Zentromere auseinander und verteilen sich im gesamten Zellkern. Wegen der dadurch hervorgerufenen Veränderung der räumlichen Ordnung im Zellkern werden nun die Zentromer-benachbarten, stillgelegten Bereiche des Erbguts (Heterochromatin) aktiviert und es häufen sich Schäden am DNA-Doppelstrang. Solche Veränderungen können die Stabilität des Genoms beinträchtigen und letztendlich zur Krebsentstehung beitragen. Für einzelne Leukämieformen ist schon bekannt, dass der Grad der Organisation des Zellkerns eine Rolle spielt. Neben NLP konnten auch Wechselwirkungen mit zwei weiteren, bereits bekannten Proteinen entschlüsselt werden. So unterstützt das Protein CTCF NLP bei der Bündelung der Zentromere. Das Nukleolusprotein Modulo wiederum verankert den Komplex aus Zentromer und NLP am Nukleolus.
Geplant sind weitere Untersuchungen, bei denen erwartet wird, dass noch andere Proteine an der Zentromer-Organisation beteiligt sind. Zudem sind wir daran interessiert, ob das aufgedeckte Netzwerk von Proteinen auch in anderen Organismen eine Rolle spielt und werden uns auf Vertreter der Nucleoplasmin Proteinfamilie in menschlichen Zellen konzentrieren.
Danksagung
PH dankt dem Human Frontiers Science Program (HFSP) für die Verleihung des Career Development Awards (CDA), der Deutschen Forschungsgemeinschaft (DFG) für die Sachbeihilfe (HE3434/2) und dem European Research Council (ERC) für die finanzielle Unterstützung durch einen Consolidator Grant (BioSynCen).

