uvCLAP
Neue Methode, um die Geheimnisse der Genregulation auf RNA-Ebene zu entschlüsseln
Unser Erbmaterial befindet sich als DNA in den Kernen unserer Zellen und muss aus dem Zellkern heraus in das Zytoplasma transportiert werden, um die genetische Information für die Herstellung von Proteinen nutzen zu können. Dazu wird die DNA zunächst in einem Prozess namens Transkription in RNA umgewandelt. Über dieses Zwischenmolekül können unsere Zellen Proteine und Enzyme produzieren.
RBP regulieren das Schicksal der RNA
Sogenannte RNA-bindende Proteine (RBP) spielen dabei eine Schlüsselrolle: Sie binden sich an die RNA im Zellkern und helfen dabei diese in das Zytoplasma der Zelle zu befördern. Darüber hinaus können RBP auch die Struktur und Funktion der gebundenen RNA-Moleküle verändern. Aufgrund dieser wichtigen biologischen Funktion, bezeichnen Wissenschaftler RBP auch als das Schweizer Taschenmesser der RNA-Prozessierung. So ist bekannt, dass verschiedene RBP ganz unterschiedliche Wirkungen auf die RNA-Moleküle haben. Während einige die Produktion von mehr Protein aus dem gleichen RNA-Molekül anregen, reduzieren andere die Proteinproduktion oder können sogar Teile der RNA herausschneiden, um nur ein bestimmtes Protein herzustellen. RBP können die Proteinsynthese aber auch verzögern, bis die RNA an eine bestimmte Stelle in der Zelle gelangt ist. Wieder andere können sogar dazu beitragen, die RNA zu zerstören, bevor sie überhaupt ein Protein produzieren kann.
Neue vielseitige Methode zur Identifizierung von Bindungsstellen von RBP
Da es im Erbgut mehr als tausend RNA-bindende Proteine gibt, ist es für die Forscher wichtig herauszufinden, welches RBP mit welchen RNA-Molekülen interagiert. So können sie besser verstehen, wie sie die Genexpression in unseren Zellen beeinflussen. Wissenschaftler des Max-Planck-Instituts für Immunbiologie und Epigenetik und der Universität Freiburg haben für genau diesen Zweck nun eine neue Technik entwickelt. Mit dieser ist es möglich, die konkreten Bindungsstellen von RNA-bindenden Proteinen an der RNA zu identifizieren.
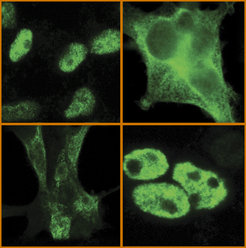
RNA-bindende Proteine können die Genexpression im Zellkern und Zytoplasma menschlicher Zellen regulieren. Hier sind drei RNA-bindende Proteine KHDRBS1 (oben, links), KHDRBS2 (unten, links) und KHDRBS3 (unten, rechts) abgebildetet, die entsprechend um den Zellkern, das Zytoplasma bzw. den Zellkern von menschlichen Zellen lokalisiert sind. Eine Punktmutation in KHDRBS1 verändert dessen zelluläre Lokalisation vollständig und verwandelt es in ein zytoplasmatisches Protein (oben, rechts). Mithilfe von uvCLAP wurden diese RNA-Bindungsstellen all dieser Proteine (und vieler weiterer) gleichzeitig untersucht, um herauszufinden, wie Änderungen in der Lokalisierung von RNA-bindenden Proteinen die Ziel-RNAs beeinflussen.
Das Verfahren mit dem Namen „Ultraviolet Crosslinking and Affinity Purification“, kurz „uvCLAP“, stellt eine schnelle, vergleichsweise einfach durchzuführende Methode zur Identifizierung von RNA-Bindungsstellen dar. Das Team um Asifa Akhtar (MPI-IE) und Rolf Backofen von der Universität Freiburg konnte zudem zeigen, dass uvCLAP sich sowohl für Fliegen-, Maus- als auch menschliche Zellen eignet. Im Gegensatz zu vergleichbaren Ansätzen kommt uvCLAP ohne gefährliche radioaktive Substanzen, lange Protokolle sowie kostenintensiven Antikörpernreagenzien aus. Durch die Verwendung von optimierten Aufreinigungs- und Multiplexing-Verfahrungen ermöglicht es dem Experimentator, kontaminierende RNA-Bruchstücke und Proteine zu entfernen und führt zu einer wesentlichen Zeitersparnis durch die gleichzeitige Verarbeitung mehrerer Proben.
Das in den letzten Jahren stark erweitere Repertoire an RNA-bindenden Proteinen sowie neuere Erkenntnisse um regulatorische Zusammenhänge in Zellen, wobei mehrere RBP um die Bindungsstellenauswahl konkurrieren oder sogar miteinander kooperieren, stellt Forscher vor immer aufwendigere und umfangreichere Experimente, um valide Daten zu erhalten. Mit uvCLAP können Wissenschaftler viele RNA-bindende Proteine gleichzeitig untersuchen und haben eine schnelle und einfach umsetzbare Methode, um die vielfältigen Protein-RNA-Wechselwirkungen in den Zellen zu studieren.
Erfahren Sie mehr Details über die uvCLAP in der Originalveröffentlichung: Die Publikation “uvCLAP is a fast and non-radioactive method to identify in vivo targets of RNA-binding proteins” veröffentlicht in Nature Communications ist frei zugänglich (open access).
