Funktion Neuronen-spezifischer 3’ UTRs
Labor Hilgers
Neuronen-spezifische ultralange 3’UTRs kommen auch beim Menschen vor; ihre Funktion ist unbekannt.
Wir gehen davon aus, dass eine ausgeglichene Expression ultralanger 3’UTRs neurogenerative Phänotypen bei Erkrankungen und normalem Altern einschränkt. Unsere Ergebnisse zeigen, dass die Mutation des Neuronen-spezifischen Teils von mRNA-Sequenzen in vivo zu schwerwiegenden neurologischen Phänotypen führen kann.
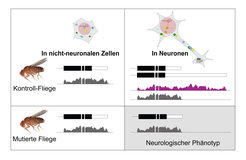
Experimentelle Strategie zur Funktionsbestimmung einzelner ultralanger 3’UTRs. Kontrollfliegen exprimieren in den meisten Zellen die kurze mRNA-Isoform, und in Neuronen die lange mRNA Isoform. Mutierten
Fliegen fehlt der neuronale Teil der 3’UTR und sie zeigen neurologische Phänotypen. mRNA-Isoformen werden schematisch mit
den zugehörigen Tracks der RNA-Sequenzierung dargestellt.
Wir wollen untersuchen, wie ultralange 3’UTRs auf die mRNA wirken, um die Gesundheit von Nervenzellen zu erhalten. Zu den Ansätzen gehören funktionale Genetik, Verhaltensanalyse, Bildgebung einzelner Moleküle, Proteomik und Transkriptomik.
