Regulierung Neuronen-spezifischer RNA-Sequenzen
Labor Hilgers
Obwohl eine typische 3’ UTR bei Drosophila einige hundert Basenpaare misst, können Neuronen-spezifische 3’ UTRs bis zu 17kb lang sein. Diese extreme Hinzufügung von Sequenz erzeugt ein erhebliches Potential für post-transkriptionelle Regulierung. Wir gehen davon aus, dass diese Extraebene an Regulierung benötigt wird, damit die mRNA eine Neuronen-spezifische Funktion erfüllt.
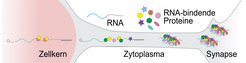
Schema der post-transkriptionellen Regulierung von mRNAs in Neuronen. RNAs sind oft mit neuronalen Granula gebündelt, die bestimmte Funktionen wie den weiträumigen Transport durchführen.
Die Rolle RNA-bindender Proteine und RNA-Kondensaten in neurologischen Pathologien, besonders neurodegenerativen Erkrankungen, wurde gut beschrieben. Ultralange 3’ UTRs könnten aufgrund ihrer ungewöhnlich hohen Zahl von Zielstellen für RNA-bindende Proteine eine Plattform für regulierten mRNA-Transport und -Translation darstellen und ein Gerüst für die Assemblierung von mRNA-Kondensaten bilden.
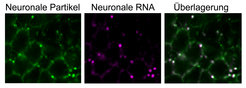
Mit Hilfe von Mikroskopie, Neuronen-Biochemie, Transkriptomanalyse und funktionaler Genetik untersuchen wir, wie neuronale RNAs, insbesondere ultralange 3’UTRs, gezielt von RNA-bindenden Proteinen reguliert werden.

